Netwokx를 활용하여 분자구조 그리기
파이썬, 그래프 라이브러리를 활용하여 네트워크 그리기
기본 이해
NetworkX는 그래프와 네트워크를 연구하기위한 Python 라이브러리로서, BSD-new 라이센스에 따라 공개 된 무료 소프트웨어이다.
이 라이브러리를 통해 그래프를 그려볼 것이다.
공식 홈페이지는 아래와 같다.
RDKit은 RDKit은 C++ 및 Python으로 작성된 화학 정보학 및 기계 학습 소프트웨어 모음이다. 이를 활용하여 화학물질을 다룰 수 있다.
공식 홈페이지는 아래와 같다.
SMILES 코드는 분자구조 화학식을 문자열로 변환하는 방법 중에 하나로 원자들의 화학 결합을 표현할 수 있다.
위키백과에 따르면 ASCII 문자열을 사용하여 화학종의 구조를 설명하기 위한 선 표기법 형식의 사양이다고 한다.

출처 : https://en.wikipedia.org/wiki/Simplified_molecular-input_line-entry_system
대상 준비
그려보고자 하는 대상은 아스피린(Aspirin) 이라는 약품이다. 이 약품의 화학식을 모르기 때문에
우선 PubChem 이라는 곳에서 Aspirin 을 검색해보면 관련 정보와 스마일스 코드를 찾을 수 있다.
PubChem은 화학 분자 및 생물학 논문에 대한 활동의 데이터베이스이다. 이 체계는 미국 국립 보건원의 일부인 미국 의학 도서관의 한 구성 요소인 미국 국립생물공학정보센터가 관리하고 있다. PumChem은 웹 사용자 인터페이스를 통해 무료로 접근할 수 있다. (위키백과 출처)
PubChem 사이트
https://pubchem.ncbi.nlm.nih.gov/
이곳에서 아스피린을 검색하여 스마일스 코드를 획득해 두도록 한다.
SMILES CODE : CC(=O)OC1=CC=CC=C1C(=O)O

활용하는 라이브러리
rdkit-pypi
networkx
matplotlib
위의 3가지 라이브러리를 pip 로 설치를 해야 한다.
RDKit으로 분자구조 그리기
필요한 라이브러리 임포트
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem.Draw import MolsToImage
from rdkit.Chem.Draw import MolToFile
import networkx as nx
import matplotlib.pyplot as plt
스마일스 코드 읽기
mol = Chem.MolFromSmiles(smiles)간단하게 한줄로 스마일스 코드를 읽어 분자 정보를 읽어 들일 수 있다.
RDKit으로 분자구조 그리기
MolsToImage(mols=[mol], subImgSize=(600,600))
간단하게 RDKit으로 아스피린이라는 물질의 분자 구조를 그려볼 수 있다.
그럼 다음에는 Networkx 로 분자구조를 그려보도록 한다.
그러기 위해서는 mol을 networkx 그래프로 전환해야 한다.
def mol_to_nx(mol):
G = nx.Graph()
for atom in mol.GetAtoms():
G.add_node(atom.GetIdx(),
labels=atom.GetSymbol(),
atomic_num=atom.GetAtomicNum(),
formal_charge=atom.GetFormalCharge(),
chiral_tag=atom.GetChiralTag(),
hybridization=atom.GetHybridization(),
num_explicit_hs=atom.GetNumExplicitHs(),
is_aromatic=atom.GetIsAromatic())
for bond in mol.GetBonds():
G.add_edge(bond.GetBeginAtomIdx(),
bond.GetEndAtomIdx(),
bond_type=bond.GetBondType(),
bond_type_value=bond.GetBondTypeAsDouble())
return G
G = mol_to_nx(mol)원자 정보는 필요한 것만 골라서 노드 정보에 담으면 된다.
이미지 그려보기
nx.draw(G)
참 쉽다.
그렇다면 노드에 레이블을 넣어 보고 싶은 마음이 생길 것이다.
nx.draw(G,pos=nx.spring_layout(G) ,with_labels = True)
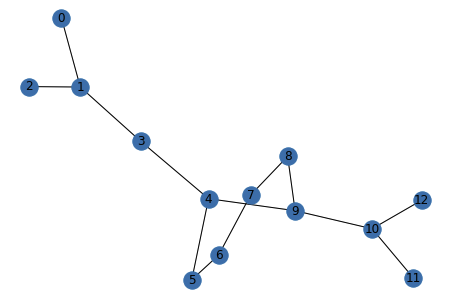
with_labels = True 설정으로 노드 id값이 기본적인 노드 레이블로 사용되어 보여진다.
참고적으로 spring_layout으로 인해 로딩할 때 마다 이미지가 변경될 것이다.
다음에는 kamada_kawai_layout 을 사용해서 레이아웃을 변경하고
노드 id 말고 위에서 노드 값에 넣어둔 심볼명을 노드 레이블로 생성하도록 하겠다.
nx.draw(G, pos=nx.kamada_kawai_layout(G), labels={node: G.nodes()[node]['labels'] for node in G.nodes()})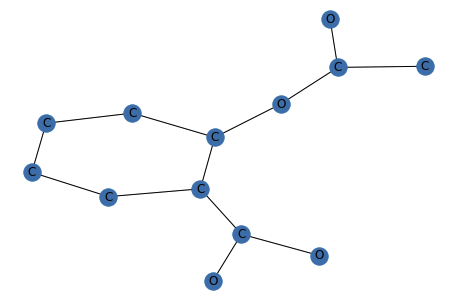
다음으로 노드의 색상도 바꾸도록 하겠다.
nColors =[G.nodes()[node]['atomic_num'] for node in G.nodes()]
color_set = list(set(nColors))
nColors = [color_set.index(n) for n in nColors]노드는 같은 원자이면 같은 색상으로 나오도록 하기 위해 원자번호를 색상 인덱스로 사용하였다.
변경하는 김에 링크에도 레이블을 추가하도록 하겠다.
plt.figure()
nx.draw(G, pos=nx.kamada_kawai_layout(G), node_color=nColors,
labels={node: G.nodes()[node]['labels'] for node in G.nodes()},
font_color='white')
nx.draw_networkx_edge_labels(
G, pos=nx.kamada_kawai_layout(G),
edge_labels=dict(((e[0], e[1]), f'{e[2]["bond_type"]}') for e in G.edges(data=True)),
font_color='red'
)
plt.axis('off')
plt.show()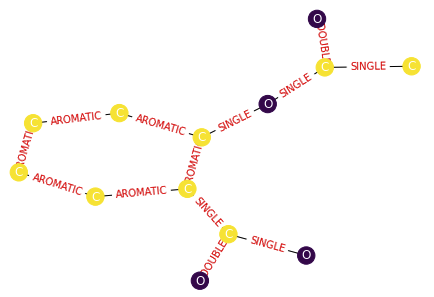
이상으로 노드의 레이블, 색상, 링크의 레이블까지 변경하여 아스피린 이라는 물질의 분자구조를 그려보았다.
SMILES 코드를 알고 있다면 위의 코드를 사용하여 손쉽게 분자구조를 그릴 수 있다.
다음 블로그 내용을 위해서
위에서 생성한 그래프의 데이터를 JSON으로 만들어 두도록 한다.
from networkx.readwrite import json_graph
origin_data = json_graph.node_link_data(G)
nColors =[G.nodes()[node]['atomic_num'] for node in G.nodes()]
color_set = list(set(nColors))
nodes=[{'id': node['id'], 'name':node['labels'], 'group':color_set.index(node['atomic_num'])} for node in origin_data['nodes']]
links = [{'source': links['source'], 'target':links['target'], 'label':str(links['bond_type']),
'value':links['bond_type_value']} for links in origin_data['links']]
gData = {"nodes":nodes,"links":links}
with open('./graphData.json','w') as f:
json.dump(gData,f, indent=4, sort_keys=True)
위의 코드로 생성되는 JSON 데이터
